Estudio de asociación de todo el genoma de tumores de la glándula mamaria en perros malteses
 Estudio de asociación de todo el genoma de tumores de la glándula mamaria en perros malteses
Estudio de asociación de todo el genoma de tumores de la glándula mamaria en perros malteses
 Keon Kim1†
Keon Kim1†  Jung Eun Canción1,2†
Jung Eun Canción1,2†  Jae Beom Joo1
Jae Beom Joo1  Parque Hyeon A1
Parque Hyeon A1  Chang Hyeon Choi1
Chang Hyeon Choi1  Chang Yun Je1
Chang Yun Je1  Ock Kyu Kim1
Ock Kyu Kim1  Parque Sin Wook1
Parque Sin Wook1  Yoon Jung Do3
Yoon Jung Do3  Tai-Young Hur3
Tai-Young Hur3  Parque Sang-Ik4*
Parque Sang-Ik4*  Chang-Min Lee1*
Chang-Min Lee1*- 1Departamento de Medicina Interna Veterinaria, Facultad de Medicina Veterinaria y Programa BK21 FOUR, Universidad Nacional de Chonnam, Gwangju, República de Corea
- 2Gwangju Animal Medical Center, Gwangju, República de Corea
- 3División de Enfermedades y Sanidad Animal, Instituto Nacional de Ciencia Animal, Administración de Desarrollo Rural, Wanju-gun, República de Corea
- 4Departamento de Patología Veterinaria, Facultad de Medicina Veterinaria y Programa BK21 FOUR, Universidad Nacional de Chonnam, Gwangju, República de Corea
Fondo: Un estudio de asociación de genoma completo (GWAS) es una herramienta valiosa para investigar la variación genética y fenotípica en muchas enfermedades.
Objetivo: El objetivo de este estudio fue identificar variaciones en los genomas de perros malteses que están asociadas con el fenotipo del tumor de la glándula mamaria (MGT) y evaluar la asociación entre cada condición biológica y el fenotipo MGT en perros malteses.
Métodos: Se extrajo ADN de 22 muestras de tumores y 11 muestras de sangre total de perros con MGT. Se realizó el genotipado de polimorfismo de nucleótido único (SNP) de todo el genoma, y los 20 SNP principales asociados con diversas afecciones y variaciones genéticas se mapearon a sus ubicaciones genéticas correspondientes.
Resultados: El proceso de genotipado identificó con éxito 173.662 loci, con una tasa general de finalización del genotipo del 99,92%. A través del análisis de control de calidad, se excluyeron 46.912 de estos SNP. Se realizaron pruebas alélicas para generar diagramas de Manhattan, que mostraron varios SNP significativos asociados con el fenotipo MGT en la región intergénica. El SNP más prominente, localizado dentro de una región asociada con la transcripción y vinculado al grado de malignidad de MGT, se identificó en el cromosoma 5 (p = 0.00001), aunque puede haber falta de significación estadística. También se encontraron otros SNP en varios genes asociados con la oncogénesis, incluidos TNFSF18, WDR3, ASIC5, STAR e IL1RAP.
Conclusión: Hasta donde sabemos, este es el primer GWAS que analiza la predisposición genética al MGT en perros malteses. A pesar del número limitado de casos, estos datos analizados podrían proporcionar la base para futuras investigaciones sobre la predisposición genética a los MGT en perros malteses.
Introducción
Los tumores de las glándulas mamarias (MGT) son la enfermedad neoplásica más común en las perras, y aproximadamente el 50% de los MGT son malignos (1-3). Se sabe que la predisposición genética, así como otros factores, como la influencia hormonal, la dieta y la obesidad, contribuyen al riesgo de desarrollar MGT (4). Las razas con un mayor riesgo de MGT pueden ser variables según cada estudio y ubicación geográfica, pero se ha informado que varias razas como malteses, caniches y yorkshire terriers tienen una mayor incidencia de MGT (5-7). La estadificación clínica de las MGT caninas se puede realizar utilizando tres factores de acuerdo con el sistema de estadificación de la Organización Mundial de la Salud: (1) evaluación del tamaño, (2) evaluación de la afectación de los ganglios linfáticos regionales y (3) identificación de metástasis a distancia (8).
Un estudio de asociación de genoma completo (GWAS) es una herramienta útil para comprender la variación genética y fenotípica. Sin embargo, su utilidad depende del nivel de desequilibrio de ligamiento (LD), ya que solo se puede genotipar un número limitado de marcadores (9, 10). En un estudio, se encontró que la LD canina era 20-50 veces más extensa que la de los humanos, lo que indica que se requieren menos SNP para el análisis de genotipos en perros que en humanos (11). Además, se estima que los perros con antecedentes de endogamia muestran bajos niveles de variación genética y un alto grado de DA dentro de las razas (10, 12). Debido a la pequeña variación genética, un origen homogéneo dentro de una sola raza puede permitir una identificación más fácil de los factores de riesgo para las MGT caninas.
Existen estudios limitados de mutaciones genéticas involucradas en el desarrollo de MGT caninos. En un estudio, se confirmó que un polimorfismo de nucleótido único (SNP) en los genes BRCA1/2, en el que se sabe que las mutaciones están asociadas con un aumento del 84% en el riesgo de cáncer de mama en medicina humana, está significativamente asociado con MGT caninos (1, 9). En otro estudio, se analizó la importancia de 10 genes asociados con el cáncer de mama humano en perros con MGT (1). Sin embargo, la mayoría de los estudios previos se han basado en mutaciones genéticas en el cáncer de mama humano.
Hasta ahora, solo un número limitado de estudios GWAS se han realizado para examinar la asociación entre SNP específicos y la susceptibilidad a MGT en perros. Melin et al. (13) fueron los primeros en realizar análisis GWAS en Springer Spaniels ingleses e identificaron genes candidatos significativos para el desarrollo de MGT canino (13). Además, más recientemente, Mucha et al. (14) realizaron un estudio preliminar de GWAS en perras con MGT, lo que llevó al descubrimiento de diferentes SNP significativos y genes candidatos en comparación con investigaciones anteriores (15). Por lo tanto, es necesario realizar análisis genéticos adicionales que incluyan otras razas a nivel de genoma completo para dilucidar los factores genéticos para el desarrollo de MGT caninos.
El propósito del presente estudio es identificar variaciones genómicas en perros malteses que afectan el desarrollo de MGT canina e investigar la asociación de polimorfismos genéticos con condiciones de MGT. Hasta donde sabemos, este estudio es el primer GWAS de MGT caninos en perros malteses.
Materiales y métodos
Animales
Treinta y tres perros malteses (22 en el grupo MGT y 11 controles) fueron incluidos en el estudio. Todos los casos y controles se seleccionaron de perros remitidos al hospital de enseñanza de medicina veterinaria de la Universidad Nacional de Chonnam y hospitales de animales locales entre julio de 2021 y junio de 2022. Los registros médicos, los exámenes físicos, las imágenes diagnósticas y los hallazgos citológicos e histopatológicos de todos los perros malteses se evaluaron retrospectivamente. Los criterios de inclusión como control incluyeron perros malteses sanos sin otras lesiones neoplásicas confirmadas por examen físico e imágenes diagnósticas. Además, se excluyeron todos los pacientes con trastornos reproductivos u hormonales. El diseño experimental del estudio fue aprobado por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Nacional de Chonnam (aprobación No. CNU IACUC-YB-2021-70).
Distribución de MGTs
La distribución macroscópica de las MGT se clasificó según si los tumores tenían una distribución bilateral (afectación de ambos lados de la cadena mamaria) o unilateral (afectación de una cadena mamaria). La evaluación de la distribución se basó en registros de exámenes físicos y fotografías de historias clínicas.
Clasificación histopatológica de los MGT
Todas las evaluaciones histológicas de MGT fueron realizadas por un solo especialista en patología. En este estudio, como condición para el análisis, los tumores mamarios se clasificaron en tumores benignos y tumores malignos.
Grado de malignidad
El grupo MGT se clasificó histopatológicamente en tumores benignos y tumores malignos, y entre los tumores malignos, los pacientes que fueron diagnosticados con carcinoma fueron calificados por el «método de clasificación de Elston y Ellis» utilizado en medicina humana (16). Los grados para el análisis de asociación fueron benignos, Grado 1, Grado 2 y Grado 3.
Obesidad
En el grupo MGT, se utilizó el sistema de puntuación de condición corporal (BCS) para distinguir entre los grupos obesos y normales (17, 18). El grupo obeso consistió en los perros con MGT con puntuaciones de 8 a 9 de 9 según el sistema BCS. El grupo normal estaba compuesto por los perros con MGT con puntuaciones de 4 a 6 de 9 en el sistema BCS.
Recolección de muestras y preparación de ADN
Después de la extirpación quirúrgica de tumores en el grupo MGT, los tejidos neoplásicos se fijaron en una solución de formalina neutralizada al 10%. El material patológico se procesó con «Pathcentre» y se incrustó en parafina. Las rodajas de 3 μm se prepararon y se tiñeron con hematoxilina y eosina (HE). Para la extracción de ADN, los tejidos neoplásicos residuales se empaparon en un tubo de microcentrífuga de 1,5 ml que contenía ARN posterior. Los tejidos empapados en ARN más tarde se almacenaron a -80 ° C hasta la extracción del ADN. Se recolectaron muestras de sangre venosa en ayunas para la extracción de ADN genómico en el grupo de control. Se añadió una alícuota de 1 ml de la muestra a un tubo anticoagulante de ácido etilendiaminotetraacético (EDTA), y los tubos se mantuvieron en hielo durante el transporte y se almacenaron a -80 ° C.
El ADN genómico se extrajo de tejidos neoplásicos y sangre entera tratada con EDTA utilizando kits comerciales (QIAamp DNA Mini Kit y QIAamp DNA Blood Mini Kit; Qiagen, Alemania) según las instrucciones del fabricante. Las concentraciones de ADN se midieron utilizando un espectrómetro. La relación A260/A280 de todas las muestras extraídas estaba dentro del rango normal de 1.7-1.9. El ADN extraído se almacenó a -20 ° C hasta el genotipado de SNP.
Estudio de genotipado y asociación de SNP en todo el genoma
El genotipado de SNPs se realizó en un laboratorio comercial utilizando el sistema BeadChip (CanineHD BeadChip; Illumina, Estados Unidos). Se analizó el ADN de 22 perros con MGT y 11 perros control. Estas muestras se procesaron durante 3 días utilizando un sistema de lectura (sistema InfiniumR HD Assay Ultra; Illumina). De acuerdo con las recomendaciones del fabricante, se realizó la preparación y cuantificación de muestras de ADN. Después de eso, las muestras se amplificaron e incubaron durante 20 h. Al día siguiente, el ADN amplificado se fragmentó mediante el uso de enzimas de restricción, y el ADN fragmentado se precipitó y se resuspendió. Las muestras de ADN preparadas se pipetearon en el BeadChip y se incubaron a 48 ° C durante 20 h para la hibridación. El día 3, las muestras hibridadas se extendieron y se tiñeron con el BeadChip. La visualización y el escaneo de las muestras teñidas se lograron utilizando una plataforma disponible comercialmente (plataforma Illumina iScan; Illumina).
Control de calidad
Software especializado (software Genome Studio; Illumina) se utilizó para analizar la salida de iScan. Las muestras de los grupos MGT y control se combinaron para optimizar la agrupación de genotipos. Los datos del genotipo polimórfico para cada grupo se analizaron por separado para evaluar los niveles de heterocigosidad y desviación del equilibrio de Hardy-Weinberg (<0,01).
Se encontró que un total de 173.662 SNP tenían polimorfismos dentro del conjunto de muestras combinadas, con más del 99% de los SNP incluidos en grupos de genotipos aceptables. En nuestras muestras, se excluyeron los SNP con tasas de llamada inferiores al 95%, los que eran monomórficos y aquellos con frecuencias alélicas menores de menos del 5% en nuestras muestras. También se excluyeron los SNP en el cromosoma X debido al desequilibrio alélico. Se realizó un análisis de componentes principales (ACP) para determinar si la distribución de las muestras de perros entre los grupos MGT y control era tendenciosa.
Asociación y análisis estadístico
Se realizaron seis análisis de asociación de 126 750 SNP mediante un programa especializado (software Variation Suite v8; Golden Helix, Estados Unidos). Se realizó un análisis estadístico de estos SNP y se consideró significativo un valor nominal de p inferior a 0,05. Las pruebas exactas de Fisher se utilizaron para comparar las proporciones de muestras con diferentes características, como fenotipo de MGT, distribución, histopatología, obesidad y esterilización o no. También utilizamos regresiones lineales multivariadas para examinar las relaciones entre diferentes grupos, incluido el grado de malignidad.
Interpretación de los resultados del GWAS
En el análisis GWAS, los valores p se obtienen probando la asociación entre cada SNP y un rasgo. Para determinar la importancia de estas asociaciones, se utiliza típicamente un nivel de significación, que a menudo se deriva de la corrección de Bonferroni, conocida como el nivel de significación de todo el genoma. Como estamos utilizando varios SNP en el análisis de GWAS, el uso de un nivel de significación de 0,05 para cada prueba individual aumentaría la probabilidad de errores de Tipo I. Para abordar este problema, la corrección de Bonferroni se realizó dividiendo el nivel de significación global por el número total de pruebas, denotado como κ. El nivel de significación ajustado (α′) después de la corrección de Bonferroni se calcula de la siguiente manera: α′ = α/κ. Aquí, α representa el nivel de significación general para todo el análisis, y κ corresponde al número de pruebas realizadas, que, en el contexto de GWAS, es equivalente al número de SNP utilizados en el análisis. En este estudio, utilizamos un nivel de significación total de 0,05, y con 126.750 SNP analizados, calculamos el nivel de significación para cada prueba, lo que resultó en un nivel de significación de todo el genoma de α′ = 3,94×10−7. Este nivel de significación ajustado se usa comúnmente en una sola prueba durante el análisis GWAS para controlar el problema de las comparaciones múltiples.
Identificación de genes y marcadores SNP
Obtuvimos datos sobre loci de SNP del navegador del genoma Ensembl (Instituto Europeo de Bioinformática, Cambridge, Reino Unido) para identificar genes asociados con SNP independientes en cada análisis. Para predecir cómo las variantes pueden afectar las transcripciones de genes, utilizamos anotaciones genéticas de la base de datos Ensembl. Nuestros hallazgos revelaron la ubicación de variantes, incluidas secuencias codificantes o intrones de transcripciones de genes empalmados alternativamente.
Resultados
Grupo de casos y condiciones de asociación
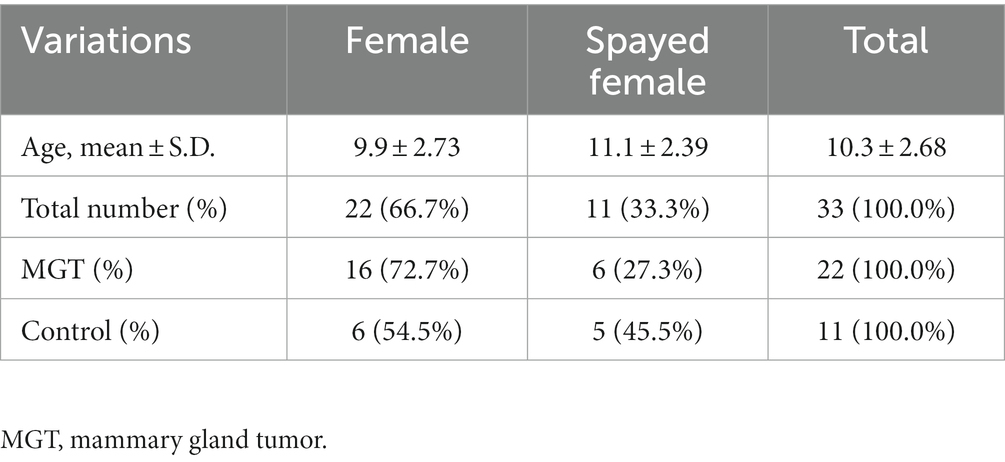
En el estudio participaron un total de 33 perras maltesas; De estos, 11 fueron esterilizados y 22 estaban intactos. La edad media era de 10,3 ± 2,72 años. La Tabla 1 resume las características fundamentales de los perros malteses. Para el estudio de asociación, se formaron seis grupos de condiciones diferentes en función de las manifestaciones clínicas de MGT, como se muestra en la Tabla 2. Un perro maltés fue excluido del análisis condicional ya que no se pudo obtener ninguna información sobre la condición asociada a MGT, excepto la castración. La evaluación histopatológica mostró que todos los tumores malignos fueron clasificados como carcinomas. El grupo benigno (n = 6) consistió en adenoma simple (n = 3), adenoma complejo (n = 1), adenoma papilar intraductal (n = 1) e hiperplasia lobulillar (n = 1). El grupo de carcinoma grado 1 (n = 6) estuvo compuesto por 4 casos de carcinoma tubular, 1 caso de carcinoma tubulopapilar y 1 caso de carcinoma in situ. Dentro del grupo de carcinoma grado 2 (n = 6), hubo 3 casos de carcinoma complejo, 1 caso de carcinoma tubulopapilar, 1 caso de carcinoma combinado con mioepitelioma maligno y 1 caso de carcinoma papilar intraductal. El grupo de carcinoma de grado 3 (n = 3) incluyó 1 caso de carcinoma tubular, 1 caso de carcinoma junto con mioepitelioma maligno y 1 caso de carcinoma complejo. Para prepararse para el análisis de variación, se extrajo ADN del tejido MGT para el grupo de casos, mientras que para el grupo de control, se extrajo ADN de leucocitos en muestras de sangre total.
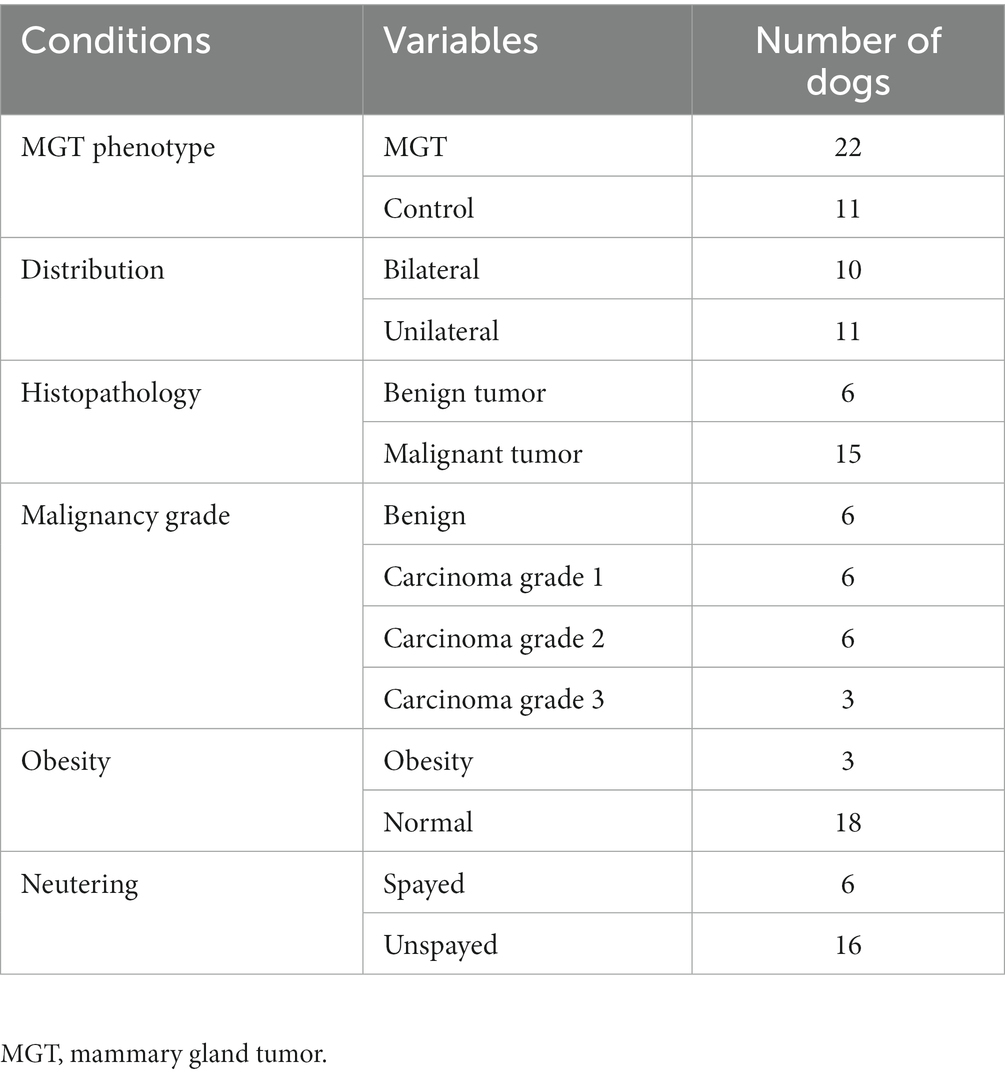 Tabla 2. Número de perros para estudios de asociación clasificados como cada condición asociada con MGT.
Tabla 2. Número de perros para estudios de asociación clasificados como cada condición asociada con MGT.
Análisis de componentes principales
El uso de PCA mostró que todos los perros incluidos en el estudio se agruparon en un grupo, como se ilustra en la Figura 1. Los resultados indican que no hubo estratificación poblacional dentro de la muestra.
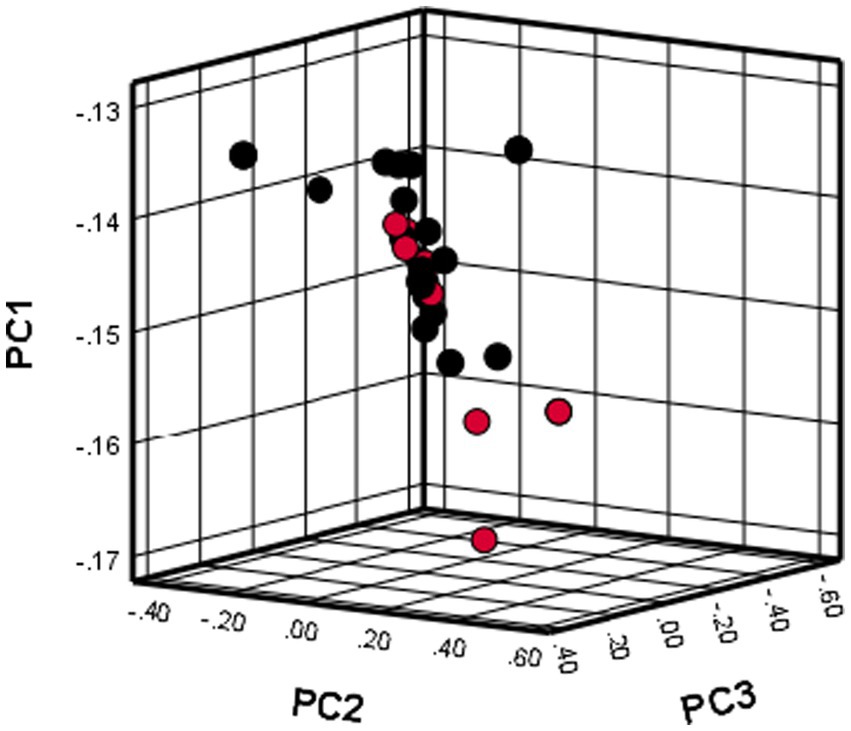 Figura 1. Resultados de la ACP que muestran la distribución de casos y controles en el presente estudio.
Figura 1. Resultados de la ACP que muestran la distribución de casos y controles en el presente estudio.
Resultados del genotipado y análisis de asociación
En este estudio, un total de 173.662 loci fueron genotipados en las muestras, y de estos, 46.912 SNPs fueron excluidos, resultando en 126.750 SNPs que fueron utilizados para el análisis posterior. La tasa general de finalización del genotipo fue del 99,92%.
Con base en los resultados del chip SNP utilizando la prueba alélica, se produjeron diagramas de Manhattan para identificar los principales loci asociados con diversas afecciones, como fenotipos de MGT, distribución macroscópica de MGT, clasificación histopatológica, grado de malignidad, obesidad y castración. La posición del mapa físico de los SNP se trazó contra los valores p −log10. No se identificaron SNP que se asociaron significativamente con el fenotipo MGT después de la corrección de Bonferroni. Sin embargo, algunos picos significativos de asociación que alcanzaron el umbral del 5% para la significación de todo el genoma (10−5) fueron identificados, como se informó en un estudio previo (19). Se encontraron tres SNP significativos asociados con el fenotipo MGT y todos se localizaron en la región intergénica (BICF2G630778783 en el cromosoma 35, BICF2P118387 en el cromosoma 34 y BICF2S23451480 en el cromosoma 21) (Figura 2). Del mismo modo, se identificaron dos SNP significativos para el grado de malignidad de MGT (BICF2P660085 en el cromosoma 18 y BICF2S2324173 en el cromosoma 11) (Figura 3). Asimismo, todos estos SNPs se localizaron en la región intergénica. Aunque puede no haber significación estadística, el SNP más significativo que se localiza en la región relacionada con la transcripción, así como asociado con el grado de malignidad de MGT se encontró en el cromosoma 5 (p = 0,00001).
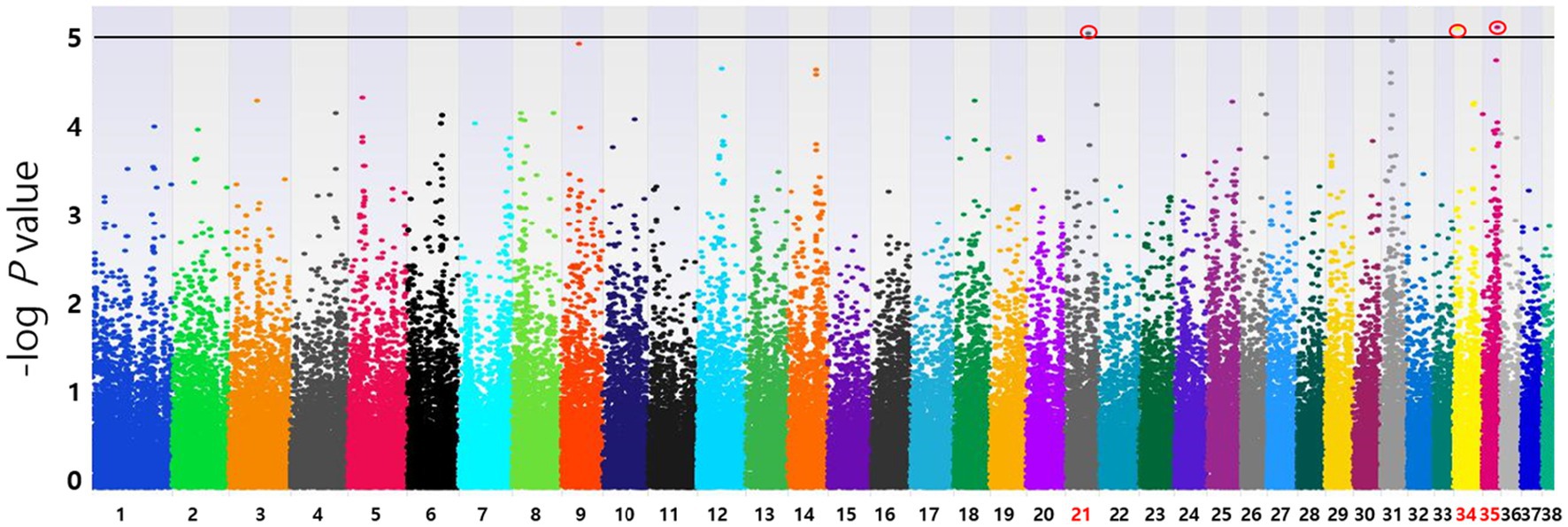 Figura 2. Diagramas de Manhattan del GWAS para el fenotipo MGT. La prueba exacta de Fisher se realizó para confirmar la significación estadística de los SNP. Los números cromosómicos se muestran en el eje X, y los resultados de cada cromosoma se muestran en el diagrama como colores individuales para cada cromosoma. El eje Y es el –log10 de los valores p calculados. Se encontraron tres SNP significativos asociados con el fenotipo MGT en el cromosoma 21(p = 9,20×10−6), 34(p = 7,96×10−6), y 35(p = 7,81×10−6). Todos los SNP estadísticamente significativos se han marcado con un círculo rojo.
Figura 2. Diagramas de Manhattan del GWAS para el fenotipo MGT. La prueba exacta de Fisher se realizó para confirmar la significación estadística de los SNP. Los números cromosómicos se muestran en el eje X, y los resultados de cada cromosoma se muestran en el diagrama como colores individuales para cada cromosoma. El eje Y es el –log10 de los valores p calculados. Se encontraron tres SNP significativos asociados con el fenotipo MGT en el cromosoma 21(p = 9,20×10−6), 34(p = 7,96×10−6), y 35(p = 7,81×10−6). Todos los SNP estadísticamente significativos se han marcado con un círculo rojo.
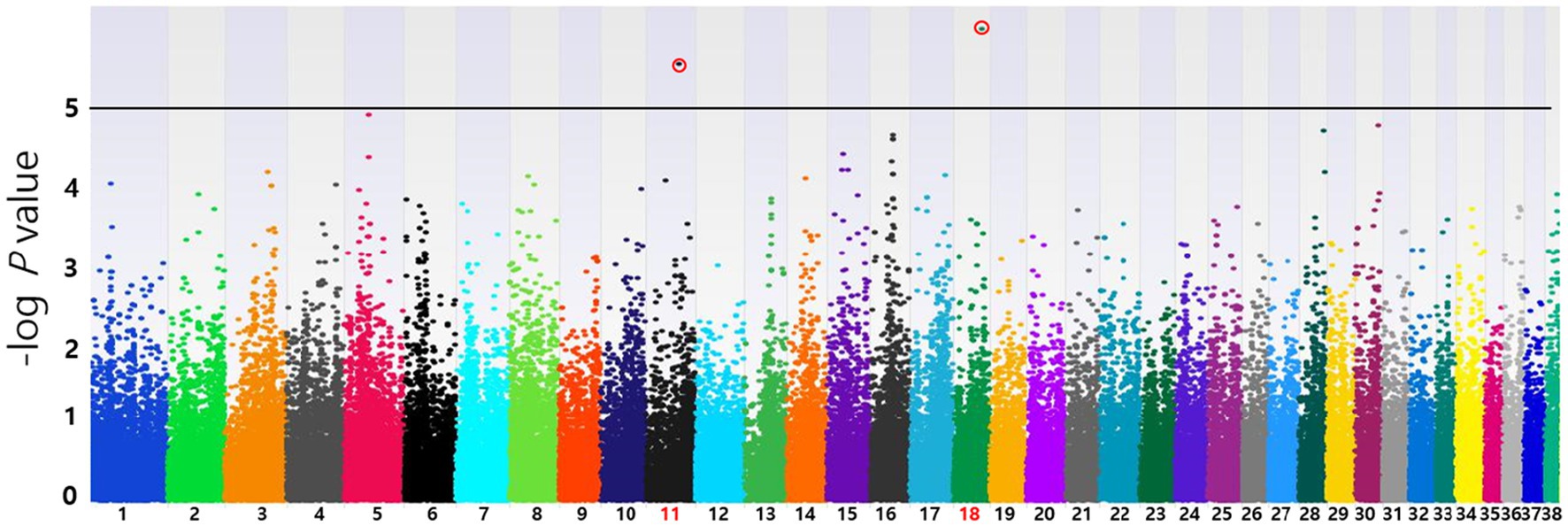 Figura 3. Diagramas de Manhattan del GWAS para el grado histopatológico de malignidad de MGT. Se realizó una prueba de regresión lineal para confirmar la significación estadística de los SNP. Los números de cromosomas se muestran en el eje X, y los resultados de cada cromosoma se muestran en el diagrama como colores individuales para cada cromosoma. El eje Y es el –log10 de los valores p calculados. Se identificaron dos SNP significativos asociados con el fenotipo MGT en el cromosoma 11(p = 2,39×10−6) y 18(p = 8,53×10−7). Todos los SNP estadísticamente significativos se han marcado con un círculo rojo.
Figura 3. Diagramas de Manhattan del GWAS para el grado histopatológico de malignidad de MGT. Se realizó una prueba de regresión lineal para confirmar la significación estadística de los SNP. Los números de cromosomas se muestran en el eje X, y los resultados de cada cromosoma se muestran en el diagrama como colores individuales para cada cromosoma. El eje Y es el –log10 de los valores p calculados. Se identificaron dos SNP significativos asociados con el fenotipo MGT en el cromosoma 11(p = 2,39×10−6) y 18(p = 8,53×10−7). Todos los SNP estadísticamente significativos se han marcado con un círculo rojo.
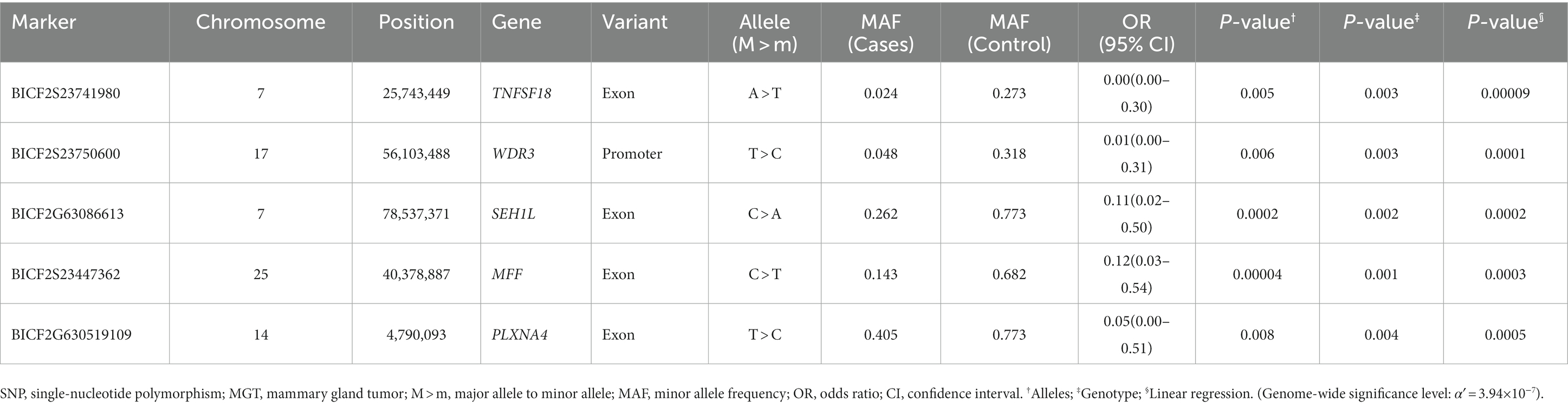
En este estudio, se identificaron los 20 SNP principales en función de sus valores de p, y se determinaron sus regiones genéticas correspondientes que se enumeran en las Tablas suplementarias S1-S6. La mayoría de los SNP para cada condición se localizaron en la región intergénica. Considerando la dificultad de dilucidar el papel de estos SNPs durante los procesos de transcripción y traducción, los hemos excluido de la tabla incluso si cumplen con los criterios de significación estadística (p < 10−5). Los SNP con una alta posibilidad de afectar la transcripción génica se resumen en las Tablas 3-5. Las Tablas 3, 4 contienen los resultados de GWAS de las pruebas exactas de Fisher para condiciones que comprenden dos grupos, mientras que la Tabla 5 presenta los resultados de GWAS de pruebas de regresión lineal para condiciones con más de dos grupos.
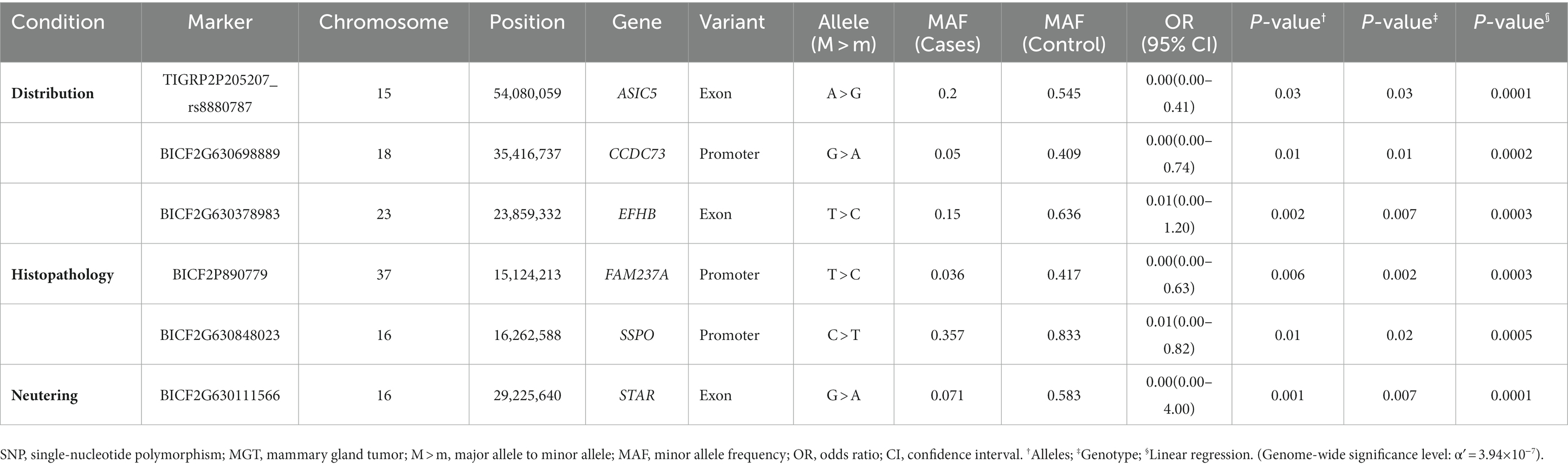 Tabla 4. Marcadores SNP candidatos asociados con diversas condiciones de MGT (distribución, histopatología y castración) derivados de las pruebas exactas de Fisher.
Tabla 4. Marcadores SNP candidatos asociados con diversas condiciones de MGT (distribución, histopatología y castración) derivados de las pruebas exactas de Fisher.
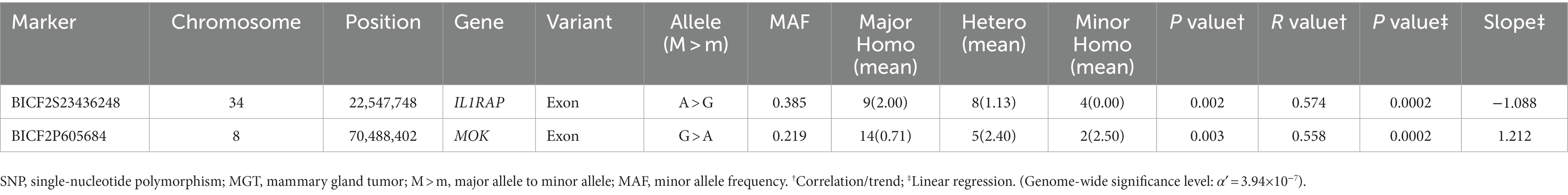 Tabla 5. Los marcadores SNP candidatos asociados con el grado de malignidad de MGT derivados de la prueba de regresión lineal.
Tabla 5. Los marcadores SNP candidatos asociados con el grado de malignidad de MGT derivados de la prueba de regresión lineal.
Discusión
Las prácticas de cría de perros de raza pura a través de la presión de selección han llevado a una disminución de la variación genética dentro de las razas (20). Debido a esta característica de los perros, están siendo reconocidos como un modelo para estudiar diversas enfermedades genéticas, e incluso un pequeño número de análisis genéticos son valiosos para evaluar sus riesgos (21, 22). En este estudio, seleccionamos perros malteses, que se sabe que son una raza con alto riesgo de MGT y son uno de los perros más comúnmente criados en Corea del Sur (5-7). Las MGT caninas y el cáncer de mama humano tienen muchas similitudes en varios aspectos. Además, dado que los perros pasan un tiempo considerable con los humanos, es muy probable que estén expuestos a los mismos factores ambientales que contribuyen a la tumorigénesis (23). Teniendo en cuenta estas características, este estudio podría establecer una base para descubrir genes candidatos que pueden afectar a los MGT en perros basados en el análisis del genoma completo, así como ampliar la investigación sobre los genes del cáncer de mama en humanos.
Se sabe que la oncogénesis de MGT es multifactorial, pero una alta incidencia de MGT en razas de perros específicas respalda un papel importante de los factores genéticos (24). Se ha informado que varios genes candidatos, como BRCA1, BRCA2, BRIP1, CHECK2, ERBB2, STK11, TOX3, TP53 y PTEN, están asociados con el riesgo de MGT canino según investigaciones previas (1, 24). Además, según el primer estudio GWAS sobre MGT canina realizado por Melin et al. (13), el gen CDK5RAP2 exhibió una asociación significativa en los Springer Spaniels ingleses (13). En este estudio, ningún SNP mostró asociación significativa (p < 10−5) con el fenotipo MGT en razas maltesas excepto para localizarse en región intergénica. Como se ve en los resultados, existe la posibilidad de la ausencia de asociaciones estadísticamente significativas con los SNP, y también existe la probabilidad de que los resultados puedan haber sido afectados por el pequeño tamaño de la muestra. Por lo tanto, será necesario un análisis adicional con un tamaño de muestra más grande en el futuro. A pesar de los resultados de que ningún SNP mostró asociación con el fenotipo MGT, el SNP más notable, situado en una región relacionada con la transcripción y vinculado al grado de malignidad de MGT, se descubrió en regiones intrónicas de genes candidatos. La participación de los intrones en la regulación de la expresión génica específica del tejido, la transcripción del ARNm y la traducción ha sido bien establecida (25). Los SNP ubicados en regiones intrón tienen el potencial de generar variantes de empalme de transcripciones y promover o interrumpir la función de los ARN largos no codificantes (lncRNAs) (26-28). En consecuencia, la susceptibilidad genética al cáncer podría verse influenciada por SNP ubicados dentro de los intrones (25).
En la condición de grado de malignidad de MGT, se encontró que genes como MAP2K4, NRG1 y PPFIA2 estaban asociados con los SNP identificados (Tabla Suplementaria S3). MAP2K4 es un miembro de la familia de activadores de la proteína quinasa activada por mitógenos (MAPK) en humanos, y se sabe que la señalización de MAPK desempeña un papel importante en la proliferación y diferenciación celular (29). En el cáncer, MAP2K4 tiene el potencial de activar la señalización de fosfoinositida-3-quinasa (PI3K) / AKT, lo que puede resultar en proliferación, migración e invasión celular. Liu et al. (30) confirmaron la posibilidad de que MAP2K4 sirva como oncogén en el cáncer de mama (30). NRG1 codifica ligandos para la tirosina quinasa del receptor heterodimérico ERBB2-ERBB3, y se cree que las fusiones génicas conducen a la estimulación del receptor autocrino (31). ERBB2 es un factor importante en el cáncer de mama en la medicina humana. Las pacientes con cáncer de mama con sobreexpresión de ERBB2 tienen tasas de supervivencia general más bajas e intervalos libres de enfermedad más cortos que las pacientes cuyo cáncer no sobreexpresa ERBB2. Además, la sobreexpresión de ERBB2 se ha relacionado con un aumento de la metástasis del cáncer de mama (32). Este estudio sugiere que los genes relacionados con la biología tumoral y el pronóstico del cáncer de mama en humanos también pueden ser aplicables a los MGT caninos. Las variaciones en los genes candidatos que están asociados con el fenotipo MGT basado en el grado del tumor podrían afectar en última instancia su expresión.
Hubo varios SNP que se encontraron en secuencias de transcripción importantes, lo que podría tener un impacto en la estructura y función de las proteínas. De estos SNP, cinco fueron particularmente significativos en relación con el fenotipo MGT y se encontraron en transcripciones de los genes TNFSF18, WDR3, SEHIL, MFF y PLXNA4 (Tabla 3). El miembro de la superfamilia TNF 18 (TNFSF18) se conoce como gen común del punto de control inmunitario. En el campo de la medicina humana, se ha demostrado que el bloqueo de los puntos de control inmunitario mejora la respuesta inmunitaria contra el cáncer, y se han identificado y aplicado clínicamente varios agentes dirigidos a los puntos de control inmunitario para tratar el cáncer. Además, Li et al. (33) confirmaron una sobreexpresión significativa de TNFSF18 en carcinomas de mama. Con base en informes anteriores, los hallazgos de este estudio sugieren que TNFSF18, identificado como un gen candidato, puede tener una anomalía de expresión asociada con variaciones de SNP en MGT canina. Esto también podría expandirse como otra opción terapéutica para MGT en perros, que actualmente no están dirigidos activamente. Se ha demostrado que el dominio de repetición WD 3 (WDR3) participa en diferentes procesos celulares, incluida la regulación génica, la transducción de señales, la progresión del ciclo celular y la apoptosis (34). Además, McMahon et al. han demostrado que la inhibición de WDR3 disminuye la proliferación de células de carcinoma de mama (35). La investigación sobre la correlación entre la sobreexpresión de WDR3 y el desarrollo de MGT no se ha realizado en los campos de la medicina humana y veterinaria, pero este estudio sugiere un área potencial para futuras investigaciones a través de sus hallazgos. La nucleoporina similar a SEH1 (SEH1L) se asocia con la glucólisis, una reacción compleja que inicia el catabolismo de la mayoría de los carbohidratos (36). Las células cancerosas se caracterizan por la reprogramación del metabolismo, y la glucólisis aeróbica es la principal fuente de energía para estas células (37). De hecho, se ha demostrado que la desregulación de la glucólisis ocurre en varios tipos de tumores (38-40).
Los SNP más significativos asociados con la distribución macroscópica incluyeron ASIC5, CCDC73 y EFHB (Tabla 4). El canal iónico sensible al ácido (ASIC) es un miembro de la subfamilia del canal degenerina/epitelial Na+ y se expresa principalmente en el sistema nervioso central y periférico (41). El microambiente tumoral es típicamente ácido debido a la vasculatura tumoral desorganizada, el flujo sanguíneo heterogéneo y el aumento de la glucólisis en las células tumorales (42). Estudios previos han demostrado que un microambiente ácido contribuye a la invasión y metástasis del tumor de mama (43–45). Además, Gupta et al. (41) sugieren que ASIC1 contribuye a la patogénesis del cáncer de mama en respuesta a microambientes tumorales ácidos (41). No obstante, dado que la función de ASIC5 sigue siendo desconocida, se necesitan más estudios para establecer las características específicas del cáncer de mama. EFHB es un regulador significativo de la entrada de calcio operada por la tienda (SOCE), un mecanismo importante que permite la entrada de calcio desde la región extracelular a través de la membrana plasmática (45). SOCE apoya una variedad de características distintivas del cáncer de mama, incluida la viabilidad celular, proliferación, migración y metástasis (45).
Se encontró que un SNP que se encontraba en el exón del gen STAR estaba entre los SNP más significativos asociados con la castración (Tabla 4). El gen STAR codifica la proteína reguladora aguda esteroidogénica (STAR), que desempeña un papel crucial en la biosíntesis de hormonas esteroides al regular el transporte de colesterol, el sustrato de todas las hormonas esteroides, desde la membrana mitocondrial externa a la interna (46, 47). En los seres humanos, se sabe que los estrógenos, particularmente el 17b-estradiol (E2), promueven el cáncer de mama (48). Además, la disfunción en la biosíntesis de andrógenos y / o estrógenos se ha relacionado con el desarrollo y crecimiento de varios cánceres que responden a las hormonas (49). Manna et al. han confirmado que la proteína STAR de las células MGT se expresa abundantemente en comparación con las células epiteliales mamarias normales y que se asocia con un aumento de los niveles de E2. Es posible que la abundante expresión de STAR facilite la entrega de colesterol a la membrana mitocondrial interna, lo que lleva a la generación de precursores adicionales para E2, lo que podría contribuir a la promoción de la tumorigénesis de mama (48).
Se sabe que la obesidad es un factor de riesgo para los efectos adversos de los tumores de mama debido a numerosas y complejas razones (50). Sin embargo, en este estudio, no se identificaron SNP significativos de acuerdo con las condiciones de obesidad en el grupo MGT. La obesidad podría verse afectada principalmente por componentes adquiridos, como los factores ambientales. Sin embargo, esto no significa necesariamente que los factores genéticos no estén involucrados. Además, sólo muy pocos casos fueron clasificados como obesos en este estudio.
En el cáncer de mama humano, se sabe que el cáncer se puede clasificar en cinco subtipos moleculares (Luminal A, Luminal B, Basal-like, ErbB2+ y Normal-like), y se ha encontrado que estas clasificaciones tienen asociaciones significativas con la supervivencia y los resultados clínicos (51). Al comparar esta clasificación de subtipo molecular en el cáncer de mama humano con la de los MGT caninos, se confirmó que comparten similitudes considerables, mientras que hay algunas diferencias (23). En ese sentido, si la probabilidad de desarrollar un subtipo específico de cáncer de mama está influenciada por factores genéticos, se anticiparía que los nuevos genes de susceptibilidad descubiertos exhiben niveles variables de expresión en cada subclase tumoral. Además, se espera que su transcripción esté regulada en cis, por SNPs dentro de ellos (52). En otras palabras, algunos de los genes candidatos asociados con SNP en posiciones cruciales para la transcripción (identificados a través del estudio GWAS) pueden exhibir diferentes expresiones en diferentes subclases de tumores. Por lo tanto, es importante recordar que en estudios futuros, los resultados de los niveles de expresión de ARNm de estos genes candidatos deben tener en cuenta los subtipos moleculares.
El presente estudio tiene varias limitaciones. Primero, el estudio tuvo un tamaño de muestra pequeño. Este tamaño de muestra limitado puede haber tenido un efecto restrictivo en la obtención de resultados estadísticamente significativos a través de la corrección de Bonferroni. Además, no se puede excluir la posibilidad de que los resultados se deban al pequeño número de muestras que conducen a la asociación significativa entre la variación genómica y el fenotipo. En segundo lugar, como el análisis GWAS para confirmar los principales SNP asociados con el fenotipo se basa en el principio de la enfermedad común de la variante común, la secuenciación de próxima generación (NGS) puede ser necesaria para un análisis riguroso del genoma completo. Finalmente, este estudio no verificó los genes candidatos identificados a nivel de traducción. En este estudio, solo las predicciones funcionales con respecto a los genes candidatos asociados con MGT se basaron en la investigación existente. Se necesitan más estudios para verificar la expresión de genes candidatos que afectan los niveles de proteínas relacionadas con la tumorigénesis.
En conclusión, este estudio examinó la correlación genética entre los SNP que están significativamente relacionados con el fenotipo MGT y varias condiciones asociadas. Se han identificado varias variaciones genéticas para el fenotipo y las condiciones de MGT, y la condición asociada más significativamente fue el grado histológico de malignidad de MGT. Hasta donde sabemos, este es el primer GWAS que analiza una predisposición genética al fenotipo MGT y las condiciones asociadas en perros malteses. A pesar del número limitado de casos, todos los datos analizados podrían ser la base para futuras investigaciones sobre la predisposición genética a los MGT en perros malteses.
Declaración de disponibilidad de datos
Los conjuntos de datos originales están disponibles en un repositorio de acceso público: Las contribuciones originales presentadas en el estudio están disponibles públicamente. Estos datos se pueden encontrar aquí: https://www.ebi.ac.uk/eva/?eva-study=PRJEB65760.
Declaración ética
Los estudios en animales fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad Nacional de Chonnam (aprobación No. CNU IACUC-YB-2021-70). Los estudios se realizaron de acuerdo con la legislación local y los requisitos institucionales. Se obtuvo el consentimiento informado por escrito de los propietarios para la participación de sus animales en este estudio.
Contribuciones del autor
KK: Conceptualización, curación de datos, análisis formal, investigación, redacción – borrador original, escritura – revisión y edición. JS: Conceptualización, Recursos, Redacción – borrador original, Redacción – revisión y edición. JJ: Curación de datos, Recursos, Redacción – borrador original. HP: Curación de datos, visualización, escritura – borrador original. CC: Curación de datos, visualización, escritura – borrador original. CJ: Curación de datos, visualización, escritura – borrador original. OK: Curación de datos, Administración de proyectos, Visualización, Redacción – borrador original. SP: Curación de datos, Administración de proyectos, Visualización, Redacción – borrador original. YD: Curación de datos, adquisición de fondos, visualización, redacción: revisión y edición. T-YH: Curación de datos, adquisición de fondos, visualización, redacción: revisión y edición. S-IP: Conceptualización, Curación de datos, Metodología, Recursos, Supervisión, Visualización, Redacción – borrador original, Redacción – revisión y edición. C-ML: Conceptualización, curación de datos, adquisición de fondos, metodología, administración de proyectos, supervisión, visualización, redacción – borrador original, redacción – revisión y edición.
Financiación
El(los) autor(es) declara(n) apoyado(s) financiero(s) fue recibido para la investigación, autoría y/o publicación de este artículo. Este trabajo fue apoyado por la Administración de Desarrollo Rural (RDA) bajo el número de subvención RS-2023-00232301 y la Agencia de Promoción de Tecnología e Información de Corea para PYMES (TIPA) bajo el número de subvención S3247434.
Reconocimientos
Los autores reconocen las valiosas contribuciones de la tarea de asesoramiento de Cheong.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Nota del editor
Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo, o reclamo que pueda ser hecho por su fabricante, no está garantizado ni respaldado por el editor.
Material complementario
El material complementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fvets.2023.1255981/full#supplementary-material
Referencias
1. Rivera, P, Melin, M, Biagi, T, Fall, T, Häggström, J, Lindblad-Toh, K, et al. El desarrollo de tumores mamarios en perros se asocia con BRCA1 y BRCA2. Cáncer Res. (2009) 69:8770–4. doi: 10.1158/0008-5472.CAN-09-1725
2. Gilbertson, S, Kurzman, I, Zachrau, R, Hurvitz, A, and Black, M. Canine mammary epithelial neoplasms: biologic implications of morphologic characteristics assessed in 232 dogs. Veterinario Pathol. (1983) 20:127–42. doi: 10.1177/030098588302000201
Resumen de PubMed | Texto completo de CrossRef | Google Académico
3. Moulton, J, Taylor, D, Dorn, C, and Andersen, A. Canine mammary tumors. Pathol Vet. (1970) 7:289–320. DOI: 10.1177/030098587000700401
4. Sorenmo, K. Tumores de la glándula mamaria canina. Veterinario Clin. (2003) 33:573–96. doi: 10.1016/S0195-5616(03)00020-2
5. Goldschmidt, MH, Peña, L, y Zappulli, V. Tumores de la glándula mamaria. En: Meuten DJ, editor. Capítulo 17: Tumores en animales domésticos. Quinta ed. (2016).
6. Kurzman, ID, y Gilbertson, SR. Factores pronósticos en tumores mamarios caninos. Semin Vet Med Surg Small Anim. (1986) 1:25–32.
7. Yamagami, T, Kobayashi, T, Takahashi, K, and Sugiyama, M. Prognosis for canine malignant mammary tumors based on TNM and histologic classification. J Vet Med Sci. (1996) 58:1079–83. DOI: 10.1292/jvms.58.11_1079
Resumen de PubMed | Texto completo de CrossRef | Google Académico
8. Owen, LN, Organization WH. TNM classification of Tumors in domestic animals. Unidad de Salud Pública Veterinaria y Centro Colaborador de la OMS para Oncolog Comparativo (1980).
9. Fesseha, H. Tumores mamarios en perros y su opción de tratamiento-una revisión. Biomed J Sci Tech Res. (2020) 30:23552–61. doi: 10.26717/BJSTR.2020.30.004980
10. Sutter, NB, Eberle, MA, Parker, HG, Pullar, BJ, Kirkness, EF, Kruglyak, L, et al. Desequilibrio de ligamiento extenso y específico de la raza en Canis familiaris. Genoma Res. (2004) 14:2388–96. DOI: 10.1101/GR.3147604
Resumen de PubMed | Texto completo de CrossRef | Google Académico
11. Ostrander, EA, y Wayne, RK. El genoma canino. Genoma Res. (2005) 15:1706–16. DOI: 10.1101/GR.3736605
Resumen de PubMed | Texto completo de CrossRef | Google Académico
12. Lindblad-Toh, K, Wade, CM, Mikkelsen, TS, Karlsson, EK, Jaffe, DB, Kamal, M, et al. Secuencia del genoma, análisis comparativo y estructura haplotípica del perro doméstico. Nature (2005) 438:803–19.
13. Melin, M, Rivera, P, Arendt, M, Elvers, I, Murén, E, Gustafson, U, et al. El análisis de todo el genoma identifica los factores de riesgo de la línea germinal asociados con los tumores mamarios caninos. PLoS Genet. (2016) 12:E1006029. doi: 10.1371/journal.pgen.1006029
Resumen de PubMed | Texto completo de CrossRef | Google Académico
14. Mucha, A, Nowak, B, Dzimira, S, Liszka, B, and Zatoń-Dobrowolska, M. Identification of SNP markers for canine mammary gland tumors in females based on a genome-wide association study – preliminary results. J. Vet Res. (2023) 67. DOI: 10.2478/JVETRES-2023-0040
15. Lindblad-Toh, K, Wade, CM, Mikkelsen, TS, Karlsson, EK, Jaffe, DB, Kamal, M, et al. Secuencia del genoma, análisis comparativo y estructura haplotípica del perro doméstico. Naturaleza. (2005) 438:803–19. doi: 10.1038/nature04338
Resumen de PubMed | Texto completo de CrossRef | Google Académico
16. Elston, CW, y Ellis, IO. Factores pronósticos patológicos en el cáncer de mama. I. el valor del grado histológico en cáncer de mama: experiencia de un gran estudio con seguimiento a largo plazo. Histopatología. (1991) 19:403–10. doi: 10.1111/j.1365-2559.1991.tb00229.x
17. Laflamme, D. Desarrollo y validación de un sistema de puntuación de condición corporal para perros. Canine Practice (1997) 22:10–15.
18. Kronfeld, DS, Donoghue, S, and Glickman, LT. Condición corporal e ingesta de energía de perros en un hospital universitario de referencia. J Nutr. (1991) 121:C157–8. doi: 10.1093/jn/121.suppl_11.S157
19. French, AT, Ogden, R, Eland, C, Hemani, G, Pong-Wong, R, Corcoran, B, et al. Análisis de todo el genoma de la enfermedad de la válvula mitral en cavalier king Charles spaniels. Veterinario J. (2012) 193:283–6. doi: 10.1016/j.tvjl.2011.09.011
Resumen de PubMed | Texto completo de CrossRef | Google Académico
20. Hedhammar, ÅA, Malm, S, and Bonnett, B. International and collaborative strategies to enhance genetic health in purebred dogs. Veterinario J. (2011) 189:189–96. doi: 10.1016/j.tvjl.2011.06.018
Resumen de PubMed | Texto completo de CrossRef | Google Académico
21. Karlsson, EK, Baranowska, I, Wade, CM, Salmon Hillbertz, NH, Zody, MC, Anderson, N, et al. Mapeo eficiente de rasgos mendelianos en perros a través de la asociación de todo el genoma. Nat Genet. (2007) 39:1321–8. doi: 10.1038/ng.2007.10
22. Karlsson, EK, y Lindblad-Toh, K. Leader of the pack: gene mapping in dogs and other model organisms. Nat Rev Genet. (2008) 9:713–25. DOI: 10.1038/NRG2382
Resumen de PubMed | Texto completo de CrossRef | Google Académico
23. Bergholtz, H, Lien, T, Lingaas, F, y Sørlie, T. Análisis comparativo del paisaje de subtipos moleculares en tumores de glándulas mamarias caninas y humanas. J Glándula Mamaria Biol Neoplasia. (2022) 27:171–83. DOI: 10.1007/S10911-022-09523-9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
24. Borge, K, Børresen-Dale, A, and Lingaas, F. Identification of genetic variation in 11 candidate genes of canine mammary tumor. Veterinario Comp Oncol. (2011) 9:241–50. doi: 10.1111/j.1476-5829.2010.00250.x
Resumen de PubMed | Texto completo de CrossRef | Google Académico
25. Deng, N, Zhou, H, Fan, H, and Yuan, Y. Single nucleotide polymorphisms and cancer susceptibility. Oncotarget. (2017) 8:110635:–110649. doi: 10.18632/oncotarget.22372
Resumen de PubMed | Texto completo de CrossRef | Google Académico
26. Pagenstecher, C, Wehner, M, Friedl, W, Rahner, N, Aretz, S, Friedrichs, N, et al. Empalme aberrante en MLH1 y MSH2 debido a variantes exónicas e intrónicas. Zumbido Genet. (2006) 119:9–22. DOI: 10.1007/S00439-005-0107-8
Resumen de PubMed | Texto completo de CrossRef | Google Académico
27. Li, L, Sun, R, Liang, Y, Pan, X, Li, Z, Bai, P, et al. Asociación entre polimorfismos en ARN largo no codificante PRNCR1 en 8q24 y riesgo de cáncer colorrectal. J Exp Clin Cancer Res. (2013) 32:1–7. DOI: 10.1186/1756-9966-32-104
28. Xiong, HY, Alipanahi, B, Lee, LJ, Bretschneider, H, Merico, D, Yuen, RK, et al. El código de empalme humano revela nuevos conocimientos sobre los determinantes genéticos de la enfermedad. Ciencia. (2015) 347:1254806. doi: 10.1126/science.1254806
Resumen de PubMed | Texto completo de CrossRef | Google Académico
29. Cuenda, A. Mitogen-activated protein kinase kinase 4 (MKK4). Int J Biochem Cell Biol. (2000) 32:581–7. doi: 10.1016/S1357-2725(00)00003-0
30. Liu, S, Huang, J, Zhang, Y, Liu, Y, Zuo, S, and Li, R. MAP2K4 interactúa con la vimentina para activar la vía PI3K/AKT y promueve la patogénesis del cáncer de mama. Envejecimiento (Albany NY). (2019) 11:10697–710. doi: 10.18632/aging.102485
Resumen de PubMed | Texto completo de CrossRef | Google Académico
31. Howarth, KD, Mirza, T, Cooke, SL, Chin, S-F, Pole, JC, Turro, E, et al. NRG1 fusions in breast cancer. Cáncer de mama Res. (2021) 23:1–12. DOI: 10.1186/S13058-020-01377-5
32. Tan, M, and Yu, D. Molecular mechanisms of erbB2-mediated breast cancer chemoresistance. Adv Exp Med Biol. (2007) 608:119–29. DOI: 10.1007/978-0-387-74039-3_9
Resumen de PubMed | Texto completo de CrossRef | Google Académico
33. Li, K, Ma, L, Sun, Y, Li, X, Ren, H, Tang, S-C, et al. El candidato a inmunoterapia TNFSF4 puede ayudar a la inducción de una respuesta inmunológica prometedora en carcinomas de mama. Sci Rep. (2021) 11:18587. DOI: 10.1038/S41598-021-98131-4
Resumen de PubMed | Texto completo de CrossRef | Google Académico
34. Su, W, Zhu, S, Chen, K, Yang, H, Tian, M, Fu, Q, et al. La sobreexpresión de WDR3 induce la activación de la vía del hipopótamo al interactuar con GATA4 en el cáncer de páncreas. J Exp Clin Cancer Res. (2021) 40:1–15. DOI: 10.1186/S13046-021-01879-W
35. McMahon, M, Ayllon, V, Panov, KI, and O’Connor, R. Ribosomal 18 S RNA processing by the IGF-I-responsive WDR3 protein is integrated with p53 function in cancer cell proliferation. J Biol Chem. (2010) 285:18309–18. DOI: 10.1074/JBC. M110.108555
36. Yu, J, Liu, T-T, Liang, L-L, Liu, J, Cai, H-Q, Zeng, J, et al. Identificación y validación de una nueva firma genética relacionada con la glucólisis para predecir el pronóstico en el cáncer de ovario. Célula cancerosa Int. (2021) 21:1–14. DOI: 10.1186/S12935-021-02045-0
37. Hanahan, D, y Weinberg, RA. Sellos distintivos del cáncer: la próxima generación. Células. (2011) 144:646–74. doi: 10.1016/j.cell.2011.02.013
38. Pavlova, NN, y Thompson, CB. Las características emergentes del metabolismo del cáncer. Celda Metab. (2016) 23:27–47. doi: 10.1016/j.cmet.2015.12.006
Resumen de PubMed | Texto completo de CrossRef | Google Académico
39. Munkley, J, y Elliott, DJ. Características distintivas de la glicosilación en el cáncer. Oncotarget. (2016) 7:35478–89. doi: 10.18632/oncotarget.8155
Resumen de PubMed | Texto completo de CrossRef | Google Académico
40. Liu, Y, Zhang, Z, Wang, J, Chen, C, Tang, X, Zhu, J, et al. La reprogramación metabólica resulta en glucólisis anormal en el cáncer gástrico: una revisión. Onco apunta a Ther. (2019) 12:1195–204. doi: 10.2147/OTT. S189687
Resumen de PubMed | Texto completo de CrossRef | Google Académico
41. Gupta, SC, Singh, R, Asters, M, Liu, J, Zhang, X, Pabbidi, MR, et al. Regulación de la tumorigénesis mamaria a través de sensores ácidos. Oncogén. (2016) 35:4102–11. DOI: 10.1038/ONC.2015.477
42. Gatenby, RA, y Gillies, RJ. ¿Por qué los cánceres tienen glucólisis aeróbica alta? Nat Rev Cáncer. (2004) 4:891–9. DOI: 10.1038/NRC1478
Resumen de PubMed | Texto completo de CrossRef | Google Académico
43. Abbey, CK, Borowsky, AD, McGoldrick, ET, Gregg, JP, Maglione, JE, Cardiff, RD, et al. Imágenes de tomografía por emisión de positrones in vivo de progresión y transformación en un modelo de ratón de neoplasia mamaria. Proc Natl Acad Sci. (2004) 101:11438–43. doi: 10.1073/pnas.0404396101
Resumen de PubMed | Texto completo de CrossRef | Google Académico
44. Gupta, SC, Singh, R, Pochampally, R, Watabe, K, y Mo, Y-Y. La acidosis promueve la invasividad de las células de cáncer de mama a través de la vía ROS-AKT-NF-κB. Oncotarget. (2014) 5:12070–82. doi: 10.18632/oncotarget.2514
Resumen de PubMed | Texto completo de CrossRef | Google Académico
45. Jardin, I, Nieto-Felipe, J, Alvarado, S, Diez-Bello, R, Lopez, JJ, Salido, GM, et al. SARAF y EFHB modulan la entrada de Ca2+ operada en almacén y son necesarios para la proliferación, migración y viabilidad celular en células de cáncer de mama. Cánceres. (2021) 13:4160. doi: 10.3390/cánceres13164160
Resumen de PubMed | Texto completo de CrossRef | Google Académico
46. Miller, WL, y Bose, HS. Primeros pasos en la esteroidogénesis: tráfico intracelular de colesterol: serie de revisión temática: genética de las enfermedades lipídicas humanas. J Lipid Res. (2011) 52:2111–35. DOI: 10.1194/JLR. R016675
Resumen de PubMed | Texto completo de CrossRef | Google Académico
47. Manna, PR, Stetson, CL, Slominski, AT, y Pruitt, K. Role of the steroidogenic acute regulatory protein in health and disease. Endocrino. (2016) 51:7–21. DOI: 10.1007/S12020-015-0715-6
48. Manna, PR, Ahmed, AU, Vartak, D, Molehin, D, and Pruitt, K. Overexpression of the steroidogenic acute regulatory protein in breast cancer: regulation by histone deacetylase inhibition. Biochem Biophys Res Commun. (2019) 509:476–82. doi: 10.1016/j.bbrc.2018.12.145
Resumen de PubMed | Texto completo de CrossRef | Google Académico
49. Chumsri, S, Howes, T, Bao, T, Sabnis, G, and Brodie, A. Aromatase, aromatase inhibitors, and breast cancer. J Esteroides Biochem Mol Biol. (2011) 125:13–22. doi: 10.1016/j.jsbmb.2011.02.001
Resumen de PubMed | Texto completo de CrossRef | Google Académico
50. Lee, K, Kruper, L, Dieli-Conwright, CM, y Mortimer, JE. El impacto de la obesidad en el diagnóstico y tratamiento del cáncer de mama. Curr Oncol Rep. (2019) 21:1–6. DOI: 10.1007/S11912-019-0787-1
51. Sørlie, T, Perou, CM, Tibshirani, R, Aas, T, Geisler, S, Johnsen, H, et al. Los patrones de expresión génica de los carcinomas de mama distinguen las subclases tumorales con implicaciones clínicas. Proc Natl Acad Sci U S A. (2001) 98:10869–74. DOI: 10.1073/PNAS.191367098
Resumen de PubMed | Texto completo de CrossRef | Google Académico
52. Kristensen, VN, y Børresen-Dale, AL. SNPs associated with molecular subtypes of breast cancer: on the utilityness of estratifica-genome-wide association studies (GWAS) in the identification of novel susceptibility loci. Mol Oncol. (2008) 2:12–5. doi: 10.1016/j.molonc.2008.02.003
Resumen de PubMed | Texto completo de CrossRef | Google Académico
Palabras clave: canino, estudio de asociación de genoma completo, maltés, tumor de glándula mamaria, polimorfismo de nucleótido único
Cita: Kim K, Song JE, Joo JB, Park HA, Choi CH, Je CY, Kim OK, Park SW, Do YJ, Hur T-Y, Park S-I y Lee C-M (2023) Estudio de asociación de todo el genoma de tumores de la glándula mamaria en perros malteses. Frente. Vet. Sci. 10:1255981. doi: 10.3389/fvets.2023.1255981
Recibido: 10 de julio de 2023; Aprobado: 19 de septiembre de 2023;
Publicado: 04 octubre 2023.
Editado por:
Naoki Miura, Universidad de Kagoshima, Japón
Revisado por:
Helga Bergholtz, Hospital Universitario de Oslo, Noruega
Md Mahfuzur Rahman, Universidad de Wisconsin–Madison, Estados Unidos
Derechos de autor © 2023 Kim, Song, Joo, Park, Choi, Je, Kim, Park, Do, Hur, Park y Lee. Este es un artículo de acceso abierto distribuido bajo los términos de la Licencia de Atribución Creative Commons (CC BY).
*Correspondencia: Parque Sang-Ik, sipark@jnu.ac.kr; Chang-Min Lee, cmlee1122@jnu.ac.kr
†Estos autores han contribuido igualmente a este trabajo y comparten la primera autoría
Renuncia: Todas las afirmaciones expresadas en este artículo son únicamente las de los autores y no representan necesariamente las de sus organizaciones afiliadas, o las del editor, los editores y los revisores. Cualquier producto que pueda ser evaluado en este artículo o reclamo que pueda ser hecho por su fabricante no está garantizado ni respaldado por el editor.
Date de alta y recibe nuestro 👉🏼 Diario Digital AXÓN INFORMAVET ONE HEALTH
Date de alta y recibe nuestro 👉🏼 Boletín Digital de Foro Agro Ganadero
Noticias animales de compañía
Noticias animales de producción
Trabajos técnicos animales de producción
Trabajos técnicos animales de compañía